Page PSMB_Seqan_2013_PEMer
Hintergrund: Paired-End Signaturen und Strukturvarianten
Als Strukturvarianten bezeichnet man üblicherweise Variante im Genom mit einer Größe von 1 Kilobasen bis 3 Megabasen [1]. Dabei betrachtet man unter anderem Insertionen, Deletionen, Translocation und Inversionen.
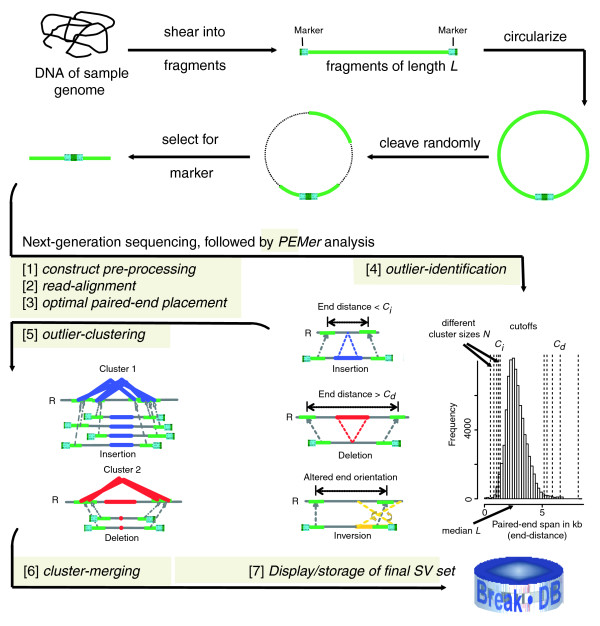
Um solche Strukturvarianten zu finden kann man sich der Information von gepaarten Reads bedienen, z.B. [2]. Dabei werden die Readpaare gegen die Referenzsequenz gemappt. Dabei entstehen bei Strukturvarianten charakteristische Signaturen. So wird z.B. der Außenabstand von Readpaaren, die um Insertionen herum gefunden werden, größer sein als derer, die an Stellen gemappt werden, die keiner Strukturvariante unterliegen.
Aufgaben
Ziel dieser Aufgabe ist es, einen Teil von PEMer nachzuimplementieren (ab Schritt 4, outlier detection). Eingabe des Programms ist eine SAM/BAM Datei mit dem Ergebnis des Read Mappings. Das fertige Programm soll dann auf simulierten Daten getestet werden. Zunächst werden Strukturvarianten in eine Referenzsequenz hineinsimuliert. Von der veränderten Referenz werden Reads simuliert und dann gegen die usprüngliche Sequenz gemappt. Das Ergebnis des Mappings wird dann in das Tool hinein gegeben. Am Ende soll die Sensitivität und die Spezifizität des Tools analog zu [2] bestimmt werden. Für die Simulation kann das Programm Mason benutzt werden, zur Simulation von Varianten gibt es in SeqAn das Programmsv_simulator benutzt werden.
Zum Mapping soll BWA benutzt werden.
Nützliche Teile von SeqAn:
- Die BamStream Klasse, siehe Basic SAM and BAM I/O Tutorial.
Die Aufgabe soll in einer kleinen Gruppe bearbeitet werden.
References
- [1] Wikipedia on Structural variation
- [2] PEMer: a computational framework with simulation-based error models for inferring genomic structural variants from massive paired-end sequencing data. Korbel, J.O., Abyzov, A., Mu, X.J., Carriero, N., Cayting, P., Zhang, Z., Snyder, M. & Gerstein, M.B. Genome Biol. 2009 Feb 23;10(2):R23
- [3] The SAM Format Speci cation (v1.4-r985)
Aufgaben Details (aus Sicht der Gruppe)
Die Folgenden Schritte gehören zur Kern-Implementierung:
- 1. spezifische Fragmentsuche nach kurzen und langen Fragmenten für Deletions und Insertions (input: SAM Dateiformat).
- 2. CLustering der gefunden Fragmente.
- 3. Bestimmung eines Thresholds zur Filterung der Clusterergebinsse.
- 4. Ergebnisse in TSV Dateiformat ausgeben.
Zusätzliche optionale Implementierungen:
- O.1. Bestimmung von (μ,σ) aus dem Inputfile.
- O.2. Automatische Coverage berechen und als Threshold setzen.
Bei den oben genannten Punkten 2 und 3 ist ein statistischer Ansatz sinnvoll und sollte bei zeitlicher Kapazität umgesetzt werden:
- S.1. Sensitivität und Spezifität (False/True Positiv/Negative) der Suche und des Clustering angeben anhand von Simulierten Daten bei denen der Erwartungswert bekannt ist.
- S.2. Vergleich der Schwankungen der Ergebnisse bei variation des Thresholds.
Stand nach dem 24.04.2013:
Kern-Implementierung:
- 1. SAM-DAtei einlesen und Fragmentsuche. (Lars Zerbe)
- 1.1. Argumentparser
- 2.1. Clustering der gefunden Fragmente. (Stephan Peter)
- 2.1.1 local Cluster
- 2.1.2 hierachical Cluster
- 2.2. Grenzwert festlegen zur Filterung. (Stephan Peter)
- 3. Ausgabe: TSV-Datei (Lars Zerbe)
optionale-Implementierungen:
- O.1. Bestimmung von (μ,σ) aus dem Inputfile.
- O.2. Automatische Coverage berechen und als Threshold setzen.
Auswertung:
- S.1. Sensitivität und Spezifität(False/True Positiv/Negative) (Lars Zerbe)
- S.2. Variieren des Grenzwerts. (Stephan Peter)
Zeitplan:
24.4 27/28.4- Entwicklung der Eingabe und der Vorverarbeitung des Input Files (Sam Datei)
- Vorbereitung des Clustering / Rücksprache
- Fertigstellung des Clustering mit fiktivem Grenzwert
- Vorbereitung der TSV Ausgabe
- Einarbeitung der optionalen Aufgaben
- Statistik-Auswertung
- Fehlerkorrektur und Feinschliff
- Ausarbeitung der Abgabe (Dokumentation etc.)
 Copyright © by the contributing authors. All material on this collaboration platform is the property of the contributing authors.
Copyright © by the contributing authors. All material on this collaboration platform is the property of the contributing authors. Ideas, requests, problems regarding Foswiki? Send feedback