Point-Pattern-Matching zur Analyse von Gelbildern
Das derzeitige Projekt geht aus einer Forschungskooperation des Instituts für Informatik der FU Berlin und des Deutschen Herzzentrums Berlin hervor. Dieses ursprüngliche Projekt wurde bis Juni 2001 von der DFG gefördert. Für Teile der dabei entwickelten Software wurde ein Lizensierungsvertrag mit der Firma Bio-Rad Laboratries abgeschlossen, der eine 2-jährige Weiterfinanzierung der Forschung und Softwareentwicklung sichert.
Gegenstand der Untersuchung sind 2-dimensionale Gelbilder, die durch Gelelektrophorese - Techniken erzeugt werden.
Die 1975 von O'Farrell eingeführte Gelelektrophorese hat sich als eine zentrale molekularbiologische Methode zur hochauflösenden Trennung von Protein-Gemischen und zur Analyse der Protein-Zusammensetzung von Gewebeproben etabliert. Jeder Punkt ("Spot") in einem so erzeugten Gelbild repräsentiert ein in der Probe auftretendes Protein. Das Original des hier verkleinert abgebildeten Herz-Ventricle-Gels enthält ca. 2000 Spots. Ziel der Analyse der Bilder ist es insbesondere, krankheitsassoziierte Proteinausprägungen zu erkennen. Zwar ist es möglich, einzelne Proteine durch Sequenzierung zu bestimmen, dies ist aber sehr teuer und aufwendig und bei der Menge der Daten nicht realistisch. Deshalb basierte bis vor wenigen Jahren die Auswertung der Gelbilder zu großen Teilen auf der genauen (und zeitaufwendigen) Betrachtung durch erfahrene Spezialisten.
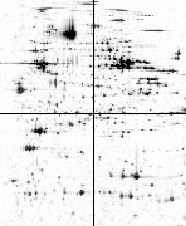
Inzwischen gibt es eine Reihe von Softwarepaketen zur Unterstützung dieser Arbeit, aber an einer hochzuverlässigen und vollautomatischen Lösung des Problems wird überall noch gearbeitet.
Im Projekt werden zwei der zentralen algorithmischen Probleme der Gelanalyse untersucht:
- Spotdetektion: Im allgemeinen konzentrieren sich die Moleküle eines Proteins aus der Probe in einer achsenparllelen elliptischen Region des Gels - dem Spot des Proteins. Bei der Spotdetektion geht es um die Erkennung dieser Regionen.Das ist eine relativ einfache Bildverarbeitungsaufgabe, so lange die Spots gut separiert sind. Wenn sich mehrere Spots zu einer komplexen und übersättigten Region überlappen, ergibt sich ein schwieriges algorithmisches Problem, das mit Approximationsalgorithmen bearbeitet wird.
- Gelmatching: Hier setzt man voraus, dass zwei zu vergleichende Bilder durch die Spotdetektion schon in geometrische Punktmuster umgewandelt wurden und nun ein geometrisches Matching dieser Punktmuster gesucht wird. Die besondere Schwierigkeit ergibt sich durch die technologisch bedingten, geometrischen Verzerrungen in den Bildern. Durch die Komplexität der Gelelektrophorese an sich, sind schon von ein und derselben Probe in einem Labor keine zwei identischen Bilder zu erwarten. Die zu entwickelnden Verfahren müssen daher sehr robust sein, um auch den Vergleich von Bildern aus verschiedenen Datenbanken zu ermöglichen. Mit Ansätzen aus der algorithmischen Geometrie konnte ein neuartiger Lösungsweg für dieses Problem entwickelt und implementiert werden, der den Kern des Programmsystem CAROL bildet (gelmatching.inf.fu-berlin.de).