Transition Path Theory of Protein:Ligand Binding Processes
Goal
Ziel: Modellierung der Bildung eines Kollisionskomplexes und des darauf folgenden Bindungvorgangs bei ausgewählten Protein:Ligand und Protein:Protein Paaren. Etablierung von TransitionPathTheoryHeld für solche Prozesse.Overview
- Einarbeitung in Theorie über metastabile Zustände, TransitionPathTheoryHeld, implizite
- Methoden: TransitionPathTheoryHeld, implicit solvent… sollen verstanden, benutzt und (soweit
- Etablierung eines methodischen Rahmens für die Bildung des
- Evtl. Implementierung auf der PS3 (falls effizient möglich)
- Untersuchung des Phänomens "superdiffusive binding".
- Simulation des Bindevorgangs aus dem Kollisionskomplex heraus mit MD und
- Möglichkeiten zur Ausweitung auf drug design abklären.
What has been done?
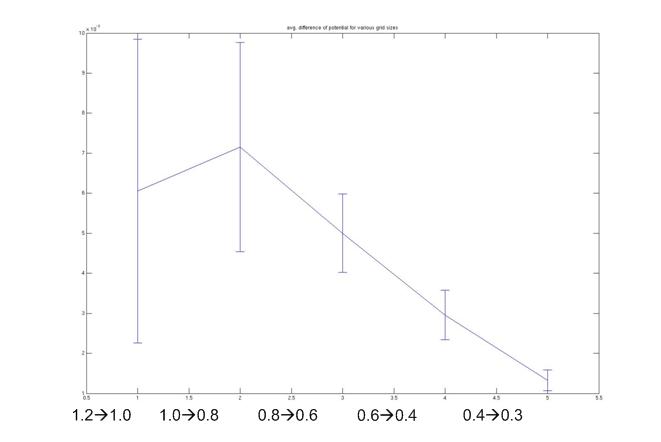
- calculation of electrotatic potential grid for 5 proteins (1MBN, 1YCQ, 3CLN, 1OLG, 1D2S) with APBS using different grid sizes (1.2, 1.0, 0.8, 0.6, 0.4, 0.3 A)
- linear interpolation of grid to next finer grid (1.2 interpolated to 1.0, 1.0 → 0.8, …)
- calculation of mean difference between interpolated grid and standard grid
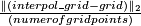
- relative distance:
Sitemap
 Copyright © by the contributing authors. All material on this collaboration platform is the property of the contributing authors.
Copyright © by the contributing authors. All material on this collaboration platform is the property of the contributing authors. Ideas, requests, problems regarding Foswiki? Send feedback